
题目
利用t-SNE模型和Procrustes分析实现基于高光谱影像的糯玉米种子分类
应用关键词
高光谱成像;糯玉米;分类;t-SNE;Procrustes分析
背景
糯玉米含有大量支链淀粉,受到世界各地消费者的青睐。品种鉴别是玉米种子质量检测的重要环节,可以防止使用假冒伪劣种子。高光谱成像(Hyperspectral imaging, HSI)技术已被应用于小麦、水稻、棉花和葡萄种子的分类,甚至利用近红外波段也可以对包衣玉米种子取得良好的品种分类结果。然而,利用HIS技术对糯玉米种子进行分类的研究很少。
首先,光谱数据数量的急剧增加,使得数据降维(Dimensionality reduction, DR)成为光谱分析中必不可少的步骤。传统的研究大多基于高光谱图像的线性假设。然而,由于像素内光谱混合、场景异质性和HSI复杂处理,使得原始光谱特征呈非线性相关。因此,有必要深入探究数据之间的非线性关系。目前,DR方法的研究主要集中在流形学习方法上,t-SNE(t-distributed stochastic neighbourhood embedding)是其中的一个研究重点。t-SNE是一种基于局部概率的非线性DR算法,它是SNE的一种变体,通过它可以减少在映射中心聚集点的趋势,从而更容易产生显著的可视化效果。已成功应用于鸟类鸣叫、计算流体动力学、基因组数据、遥感图像等分析中。
其次,现有的大多数图像分析方法都假设训练集和测试集处于完全相同的实验条件和数据分布中,尽管这种假设在大多数情况下是无效的,因为即使在相同的条件下,光线或位置也会发生变化。因此,数据之间的漂移是需要考虑的问题。Procrustes分析(PA)是一种通过匹配一个集到另一个集的数据变量相关性来解决这一问题的方法。通过连续迭代将形状差异的度量最小化,PA可以有效消除数据移动的影响。本研究将其作为一种预处理方法应用于高光谱图像处理中。
最后,将目前广泛应用的Fisher判别分析(FDA)应用于糯玉米分类。在t-SNE方法获得局部判别信息后,通过最大化类间散射和最小化类内散射来补充全局判别信息。
本研究旨在探讨基于高光谱影像的PA和t-SNE技术在糯玉米种子品种分类中的可行性。具体目的是(1)比较基于t-SNE、PCA、KPCA和LLE的分类模型的性能;(2)利用PA算法对数据进行预处理,提高分类精度;(3)比较玉米种子胚侧和非胚侧高光谱图像的差异。
试验设计
仲恺农业工程学院唐宇教授团队利用可见/近红外高光谱成像系统(GaiaSky-mini, 江苏双利合谱公司)获取了800粒玉米籽粒的侧胚以及非侧胚影像。该成像系统光谱范围为386.7-1016.7 nm,有256个波段,包括一个CCD相机(ICX285, 索尼),两个用于照明的50 w LED灯,以及由步进电机驱动的传送带(ZOLIX SC300, 卓立汉光)。
首先,获取每粒种子的两侧胚和非胚部分的高光谱影像(图1a);其次,选取感兴趣区域并计算其平均光谱(图1b);然后对光谱进行标定(图1c);采用PA算法对光谱数据进行预处理(图1d);最后,利用t-SNE等模型进行种子品种分类(图1e)。
为了减少数据冗余,研究中使用连续投影算法SPA来选择最佳波长,并使用交叉验证中的均方根误差来评估模型性能。具体训练与测试模型的过程如图2所示。
为了比较有无PA预处理的t-SNE、PCA、KPCA和LLE分类模型的效果,本研究共建立了8个比较组合(PCA+FDA、KPCA+FDA、LLE+FDA、t-SNE+FDA、PA+PCA+FDA、PA+KPCA+FDA、PA+LLE+FDA、PA+t-SNE+FDA)。
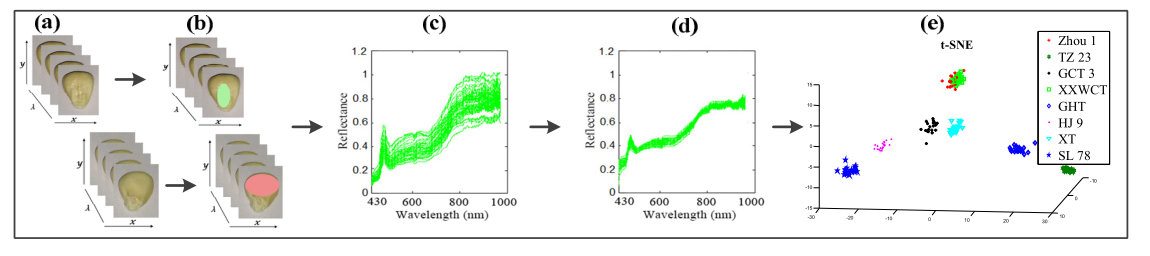
图1 糯玉米种子品种分类图像处理及数据分析流程图。
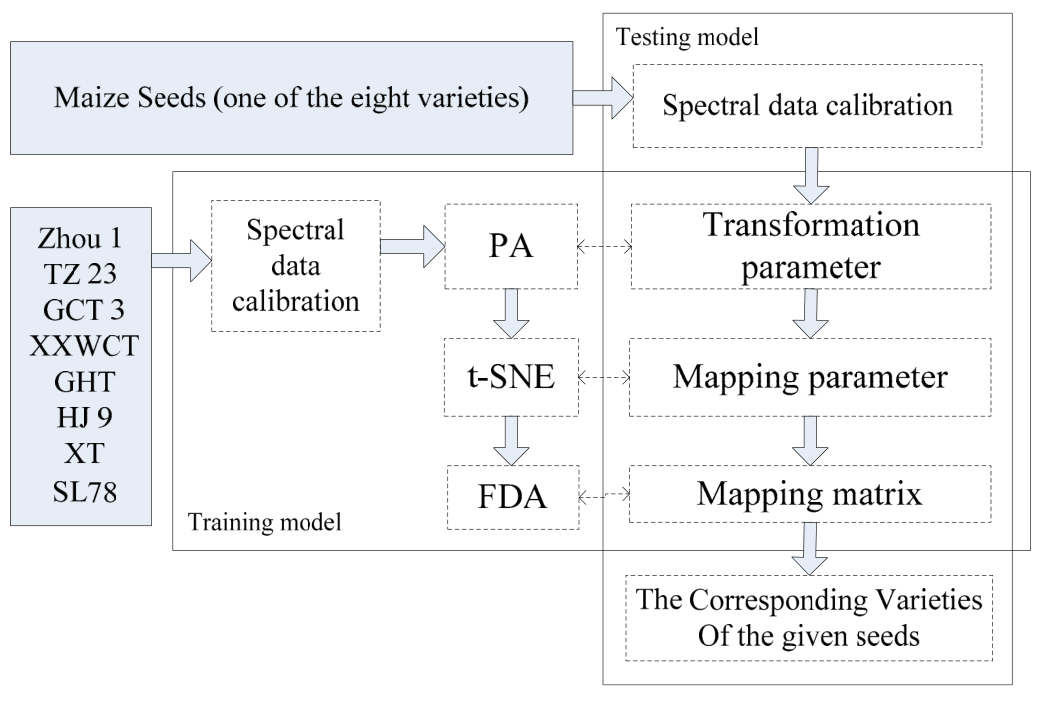
图2 训练与测试模型的详细过程
结论
利用SPA为非侧胚和侧胚选择的最佳波段分别为455.5 nm、697.6 nm、495.1 nm、841.2 nm、732.4 nm、653.3 nm、887.7 nm、833.6 nm和737.4 nm、578.1 nm、460.2 nm、932 nm、937.2 nm、945.1 nm、947.7 nm、960.9 nm。非侧胚和侧胚选择的波段有较大差异是因为胚胎侧含有大量淀粉、油和其他化合物。
虽然在分析前对高光谱图像进行了校正,但噪声仍然存在(图3a)。对于同一玉米品种,由于光照和噪声的影响,所有的高光谱图像在光谱上沿波段散射。在这种情况下,不同类别的数据可能会重叠,最终也会影响分类模型。相同品种种子的高光谱图像的分布可能具有相同的变异和最小的偏差。PA算法的目的就是减小数据方差,使高光谱数据的差异最小化。该算法具有良好的性能,适用于原始光谱的预处理。得到的预处理光谱曲线如图3b所示。可以看出,PA预处理后的光谱更加聚集,重叠的数据变少,并且增强了原始光谱的信息。
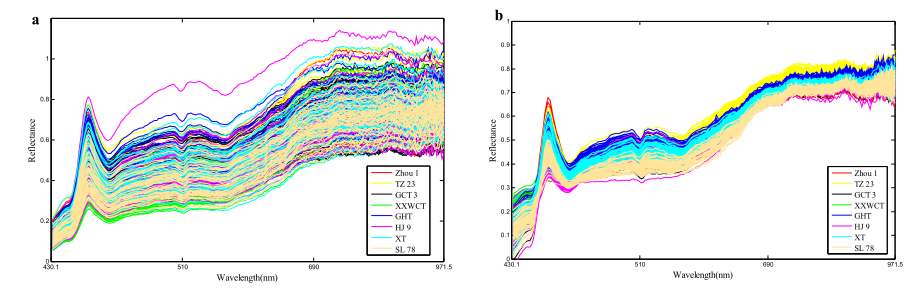
图3 原始光谱曲线(a)以及经过PA处理后的光谱曲线(b)
8个玉米品种降维后的三维分布如图4所示。对于PCA,不同品种之间没有明显的分离,同一类样品沿空间分布并重叠。LLE的散点图与PCA的散点图基本相同。KPCA的区分效果优于PCA和LLE,但较t-SNE差。t-SNE表现优异,品种TZ23、GCT3、GHT、HJ9、XT和SL78的分布和区域有明显区别,而Zhou1和XXWCT在同一分布区域并有一定的重叠。这是由于t-SNE在捕获局部数据特征和区分数据结构方面有一定优势。具有相似几何结构的糯玉米种子的高光谱图像基本是相似的,图中相邻的光谱曲线具有高度的相关性。因此,利用光谱曲线的局部特征可以更容易地表示数据的变化。
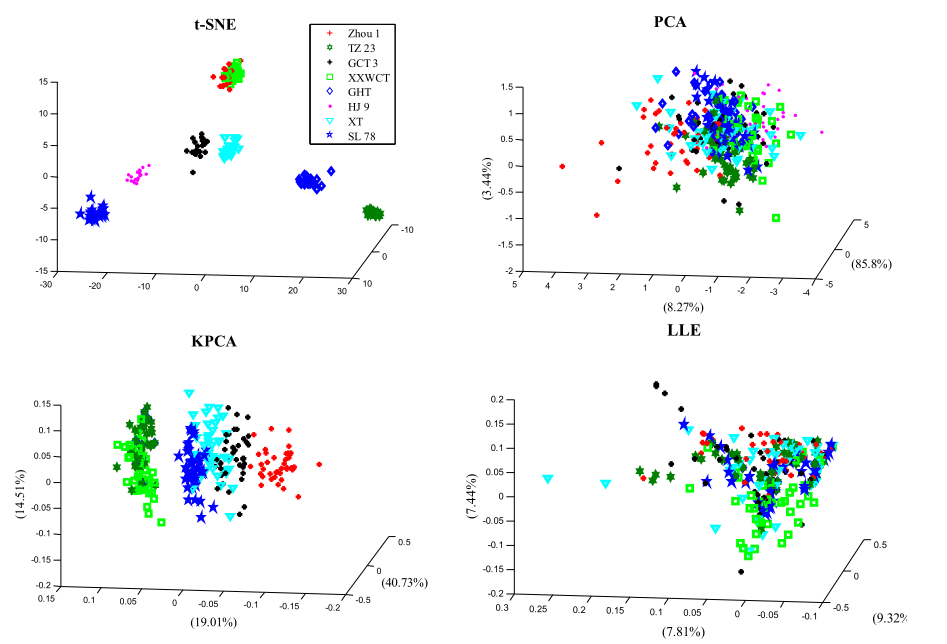
图4 基于不同降维方法的样本三维散点图
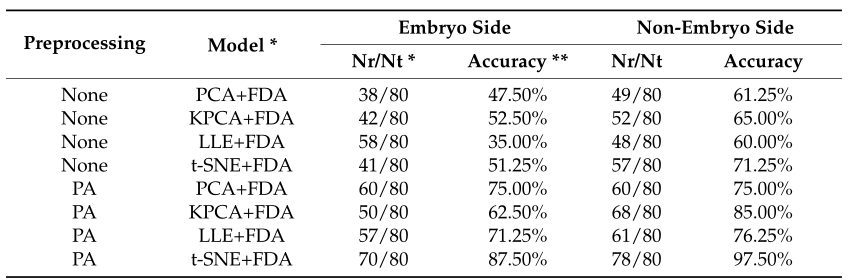
在对光谱进行不同的处理方式后,使用FDA算法对玉米种子品种分类,分类结果如表1所示。对于侧胚,PCA+FDA、KPCA+FDA、LLE+FDA和t-SNE+FDA模型的准确率接近50%,采用PA将模型的准确率提高到62.5~87.5%。对于非侧胚,所有的准确率都超过60%。
对于不含PA的模型,测试准确率仅为35~71.25%。相比之下,PA模型具有更好的结果,准确率为62.5~97.5%。PA效果显著可能由于它是一种聚集效应,这已被证明有助于不同领域的分类。无PA预处理时,KPCA与t-SNE具有相似的分类精度,特别是对侧胚数据,但准确率低于有PA预处理的t-SNE。这一结果表明了核函数选择在KPCA中的不稳定性和重要性。作为一种基于核的算法,不同的选择对KPCA的性能影响很大,很难给出稳定、鲁棒的分类结果。
t-SNE模型的准确率高于其他模型,其中PA预处理的t-SNE模型在所有方法中效果最好。使用非侧胚数据的准确率为97.5%,其他模型的最佳准确率仅为85%。总体而言,除KPCA+FDA模型,t-SNE模型在进行或不进行PA预处理的情况下,模型性能均为最优。
表1 不同模型的分类结果
作者信息
唐宇,博士,仲恺农业工程学院教授,博士生导师。
主要研究方向:农业电气化与自动化等。
参考文献:
Miao, A., Zhuang, J., Tang, Y., He, Y., Chu, X., & Luo, S. (2018). Hyperspectral Image-Based Variety Classification of Waxy Maize Seeds by the t-SNE Model and Procrustes Analysis. Sensors (Basel), 18. https://doi.org/10.3390/s18124391
 地址:无锡市梁溪区南湖大道飞宏路58-1-108
地址:无锡市梁溪区南湖大道飞宏路58-1-108
.png) 电话:13810664973
电话:13810664973
.png) 邮箱:info@dualix.com.cn
邮箱:info@dualix.com.cn
 地址:北京市海淀区中关村大街19号
地址:北京市海淀区中关村大街19号
.png) 电话:13810664973
电话:13810664973
.png) 邮箱:info@dualix.com.cn
邮箱:info@dualix.com.cn
 地址:陕西省西安市高新区科技一路40号盛方科技园B座三层东区
地址:陕西省西安市高新区科技一路40号盛方科技园B座三层东区
.png) 电话:13810664973
电话:13810664973
.png) 邮箱:info@dualix.com.cn
邮箱:info@dualix.com.cn
 地址:成都市青羊区顺城大街206号四川国际大厦七楼G座
地址:成都市青羊区顺城大街206号四川国际大厦七楼G座
.png) 电话:13810664973
电话:13810664973
.png) 邮箱:info@dualix.com.cn
邮箱:info@dualix.com.cn
 地址:深圳市龙华区民治梅龙路
地址:深圳市龙华区民治梅龙路.png) 电话:13810664973
电话:13810664973.png) 邮箱:info@dualix.com.cn
邮箱:info@dualix.com.cn